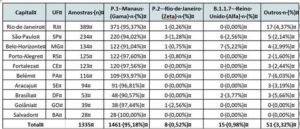
Segundo dados do Grupo Pardini, em mais de 95% das amostras avaliadas em todas as capitais entre abril e maio de 2021, predomina a variante P.1 – Manaus. Outros 3% representam possíveis novas variantes ainda não descritas. A preocupação agora é com a variante Delta que, em breve entrará no monitoramento.
O surgimento de novas variantes de SARS-CoV-2 tem agravado cada vez mais o cenário da COVID-19 no Brasil e no mundo e trouxe a necessidade de uma vigilância genômica constante. Por isso, o Grupo Pardini, por meio de sua equipe de Pesquisa e Desenvolvimento (P&D) em colaboração com o Laboratório de Biologia Integrativa do ICB/UFMG, desenvolveu um sistema de vigilância epidemiológica para identificar a ocorrência das principais variantes presentes no Brasil desde abril de 2021, com cobertura nacional e capacidade de detecção de novas variantes com frequência de até 5% na população.
A iniciativa é mais uma comprovação prática da contribuição do Grupo Pardini para o combate à pandemia no Brasil. “Entendemos que o patamar em que se encontra a Covid-19 no Brasil, requer urgência de esforços de vigilância genômica em todo o país, para a avaliação da situação destas novas variantes de SARS-CoV-2. Visando utilidade pública, vamos dar publicidade às informações sobre a incidência das variantes no país, de forma prática, para apoiar futuras decisões sociais, políticas, econômicas e claro, dos órgãos oficiais de saúde”, explicou o vice-presidente da rede de laboratórios, Alessandro Ferreira.
Os resultados do estudo já realizado em abril e maio de 2021 mostram uma predominância da variante P.1 – Manaus (Gama) em mais de 95% das amostras avaliadas, seguida das variantes B.1.1.7 Reino Unido (Alfa) e P.2 – Rio de Janeiro (Zeta) que representaram 0,98% e 0,52%, respectivamente. A variante B.1.351 (África do Sul) não foi detectada nesta amostragem.
Além disso, curiosamente, 3% das amostras avaliadas representam possíveis novas variantes de SARS-CoV-2, uma vez que apresentaram comportamento anômalo na genotipagem. Elas serão reavaliadas por sequenciamento do genoma viral para a identificação de variantes ainda não descritas.
Para o vice-presidente do Grupo Pardini, Alessandro Ferreira, um dos grandes desafios frente ao controle da pandemia da Covid-19 é o rastreamento efetivo e em tempo real de variantes. “Estas informações podem orientar principalmente ações de saúde pública a fim de conter a transmissão de novas variantes, além de subsidiar pesquisas que buscam relacionar as alterações na capacidade de transmissão, incidência de casos graves e direcionar estratégias de vacinação para regiões mais críticas e com predominância de variantes mais transmissíveis”, afirma o geneticista.
Este estudo com a incidência das variantes nas principais capitais do Brasil será divulgado quinzenalmente, no Boletim Pardini Covid-19, junto com a atualização dos dados epidemiológicos. A identificação destas tem sido tratada com grande atenção pela comunidade científica e autoridades de saúde e o Grupo está constantemente monitorando a dinâmica das variantes em território nacional, em tempo real, através da capilaridade e cobertura nacional da rede de laboratórios do instituto.
Até o momento, o monitoramento em andamento, analisou 1335 amostras distribuídas pelas capitais: Rio de Janeiro (389), São Paulo (234), Belo Horizonte (134), Porto Alegre (125), Fortaleza (123), Belém (116), Aracaju (94), Brasília (53), Goiânia (39), Salvador (28). As variantes de SARS-CoV-2 foram identificadas através da genotipagem por PCR em tempo real para as mutações N501Y, K417T, K417N e E484K no gene da espícula viral capaz de caracterizar as variantes: B.1.1.7 Reino Unido (Alfa); B.1.351 África do Sul (Beta); P.1 – Manaus (Gama) e P.2 – Rio de Janeiro (Zeta).
“O objetivo do Laboratório Observacional de COVID-19 do Grupo Pardini é realizar o monitoramento e vigilância genômica de SARS-CoV-2 em tempo real, com amostragens periódicas (intervalos de 15 dias) em todo território brasileiro. Esta ação reforça a cultura inovadora da companhia e seu compromisso com a sociedade e a ciência e a geração de dados para o enfrentamento da COVID-19”, acrescentou Alessandro Ferreira.
Dados da vigilância genômica do Laboratório Observacional do Grupo Pardini